La dégradation des ARN est essentielle à la régulation de l’expression des génomes. Diverses voies de dégradation des ARN coopèrent pour assurer :
- l’élimination des ARN codants et non-codants en réponse aux signaux développementaux et environnementaux
- l’élimination de transcrits défectueux (Contrôle de qualité des ARN)
- la dégradation de transcrits cryptiques produits à partir de régions intergéniques (Surveillance des ARN)
- la lutte contre des pathogènes comme les virus.
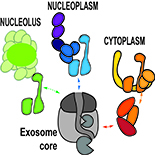
Les objectifs principaux de notre équipe sont d’identifier des acteurs clés des voies de dégradation des ARN et de déterminer leurs rôles dans la régulation de l’expression des gènes, leur impact sur le développement ou la résistance aux stress des plantes. Nous sommes particulièrement intéressés par les co-facteurs de l’exosome, par les enzymes responsables de l’adénylation et l’uridylation des ARN, et par de nouveaux facteurs associés aux P-bodies et aux activateurs du décapping. Enfin, nous évaluons l’importance des voies de dégradation des ARN au cours d’infections virales.
Nos projets intègrent des stratégies de génétique directe et inverse chez Arabidopsis thaliana, de purification des protéines couplée à des analyses en spectrométrie de masse, ainsi que des protocoles spécifiques de séquençage à haut débit (Illumina et Nanopore) pour caractériser les extrémités 3’ des ARN.
Notre programme de recherche est actuellement financé par le LabEx NetRNA (2011-2028) et les contrats ANR URIVir (2021-2025) et SeedUTail (2022-2026).