Les protéines à « pentatricopeptide repeat » (PPR) forment une classe majeure de protéines de liaison à l’ARN universellement conservée chez les eucaryotes. Ces protéines sont impliquées dans des processus d’expression génétique essentiels principalement dans les organites.
Nous étudions des protéines PPR impliquées dans les processus post-transcriptionnels et dans la traduction chez les mitochondries de plantes en combinant des approches biochimiques, structurales et de génétique inverse. .
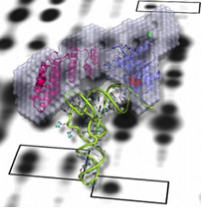
Nous avons notamment identifié des protéines PPR appelées PRORP responsables de l’activité RNase P (maturation en 5’ des précurseurs d’ARNt). Nous cherchons à identifier les différents substrats et les partenaires protéiques des PRORP. La caractérisation fonctionnelle des protéines PRORP chez d’autres eucaryotes nous permet d’appréhender l’évolution de cette enzyme. En plus du domaine PPR, les protéines PRORP contiennent un domaine catalytique de type NYN. Nous nous intéressons à la diversité fonctionnelle des nucléases NYN chez les plantes.
Nous avons aussi identifié des protéines PPR qui sont parties intégrantes du mitoribosome et donc impliquées dans la traduction mitochondriale des plantes.
Les recherches menées dans l’équipe permettront de comprendre les fonctions moléculaires de ces protéines et leur rôle biologique à l’échelle de la plante. Les résultats obtenus ouvrent un large champ d’applications allant de l’amélioration des plantes à la santé humaine.
L’équipe fait partie du consortium LabEx MitoCross. Elle est également soutenue par plusieurs financements ANR.