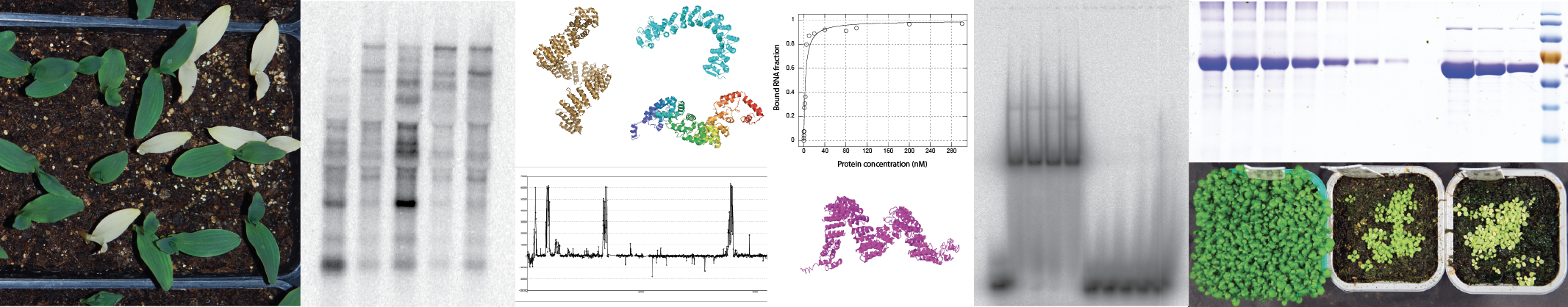
Le chloroplaste joue un rôle pivot dans la régulation de l’homéostasie de la cellule végétale de par son rôle de senseur de la lumière et de production de métabolites essentiels à la cellule. La biogénèse et le fonctionnement de cet organite repose sur l’expression concertée de son génome d’origine bactérienne et de gènes portés par le génome nucléaire. La régulation de l’expression génétique du chloroplaste est primordiale à l’adaptation de la plante à son environnement et durant son développement. Dans les chloroplastes, les étapes post-transcriptionnelles de l’expression génétique sont particulièrement complexes et requièrent de nombreuses protéines de liaison à l’ARN qui sont codées par des gènes nucléaires dont seule une petite fraction a été identifiée. Ces protéines qui sont importées dans l’organite contrôlent virtuellement l’ensemble des processus nécessaires à la maturation des ARNs chloroplastiques.
Notre équipe de recherche combinent des approches de génétique inverse chez Arabidopsis et le maïs, de biologie moléculaire et de biochimie des complexes ribonucléoprotéiques afin d’identifier et résoudre les fonctions de ces protéines dans les chloroplastes. Nous visons également à comprendre comment ces protéines de liaison à l’ARN participent à l’adaptation génétique du chloroplaste en réponse aux changements environnementaux en identifiant les voies de signalisation cellulaire permettant de réguler leur activité intrinsèque, in vivo.