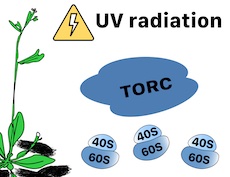
L’exposition au stress génotoxique (UV) peut affecter l’intégrité cellulaire et endommager les compartiments nucléaire (ADN) et cytoplasmique (ARN), altérant l’intégrité de l’(épi)génome et l’expression des gènes (transcription et traduction). Ainsi, la capacité des organismes vivants à faire face aux effets délétères des agents génotoxiques repose sur les interactions entre différents processus de surveillance empêchant les réarrangements chromosomiques, les altérations de l’épigénome et la baisse d’activité de la machinerie transcriptionnelle/traductionnelle. Nous souhaitons décrypter les mécanismes moléculaires contrôlant l’intégrité de l’(épi)génome et la traduction en réponse à une exposition aux stress génotoxiques.
Surveillance de l’(épi)génome