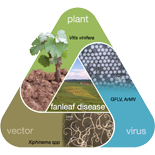
De nombreux virus figurent parmi les pathogènes de la vigne les plus dommageables et les plus répandus mondialement (court-noué, enroulement, etc…). Située à l’interface entre recherche fondamentale et appliquée, l’objectif global du groupe « Biologie et Biotechnologie des virus de la vigne », est de mieux connaître la biologie de ces virus afin de comprendre leurs interactions avec la plante hôtes, la manière dont ils se propagent de cellule en cellules via les plasmodesmes et leur mécanisme de transmission par vecteur. Ceci nous permet de proposer de nouveaux outils ou de nouvelles stratégies pour lutter contre ces agents : détection et phytodiagnostic à l’aide de Nanobodies, obtention de plantes résistantes, biotechnologies dérivées de Virus-Like-Particles (VLP), etc…, et ainsi de renforcer notre position parmi les leaders internationaux en virologie et biotechnologie des virus de la vigne.
Développement de Nanobodies contre les principaux virus de la vigne
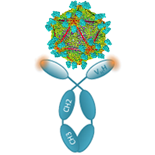
Les Nanobodies (Nb) sont de petits peptides dérivés d’anticorps simple-chaine trouvés chez les camélidés. Découverts dans les années 90, les Nb sont les plus petites molécules de type anticorps connues et suscitent un vif intérêt en biotechnologie. Nous avons montré que les Nb dirigés contre le Grapevine fanleaf virus (GFLV) possèdent une activité antivirale et confèrent à la plante une résistance au virus. Ils peuvent servir pour l’immunodétection du virus et sont aussi d’excellents biocapteurs du GFLV in planta. Sur le même principe et dans le même but, nous développons des Nb contre d’autres virus majeurs de la vigne responsables des maladies du court-noué (nepovirus), de l’enroulement (ampelovirus) et du complexe du bois strié (vitivirus).