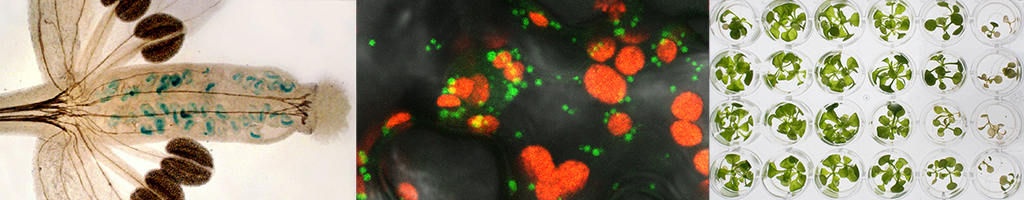
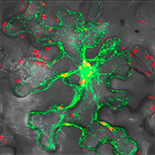
Notre équipe s’intéresse aux mécanismes qui contrôlent la maintenance, la ségrégation et la réparation du génome mitochondrial (mtDNA) des plantes, ainsi qu’aux processus de régulation antérograde (noyau vers les organelles) et rétrograde (organelles vers le noyau) qui gèrent son expression.
Les mitochondries des plantes supérieures possèdent des génomes de grande taille avec une structure dynamique résultant d’événements de recombinaison. Ces mécanismes de recombinaison sont nécessaires pour la réplication et la ségrégation stœchiométrique de l’information génétique mitochondriale. Ils sont aussi responsables de l’évolution très rapide de la structure du mtDNA. De plus, la maintenance de ce génome complexe dans l’environnement oxydatif généré par l’activité de la chaîne respiratoire requiert des mécanismes efficaces de réparation de l’ADN. L’étude de ces mécanismes reste peu développée. Nous nous intéressons plus particulièrement à la réparation recombinaison-dépendante mais aussi à la réparation par excision de base (« BER » pour « Base Excision Repair ») ou par excision de nucléotide (« NER » pour « Nucleotide Excision Repair »). Nous étudions ces voies et les facteurs impliqués, en utilisant les outils génétiques, génomiques, biochimiques et de biologie cellulaire disponibles pour la plante-modèle Arabidopsis.
Pour l’étude des processus de régulation génétique mitochondriale et de coordination avec le noyau, nous avons développé une stratégie originale permettant d’invalider de façon inductible des ARN mitochondriaux individuels. Cette approche nous permet d’analyser la dynamique de la réponse à un changement brutal dans l’équilibre normal du transcriptome mitochondrial, tandis qu’un mutant classique serait au contraire représentatif d’une nouvelle situation d’équilibre.