La plateforme est spécialisée dans le séquençage des acides nucléiques et l’analyse de l’expression des gènes. Stratégiquement, elle constitue un pôle d’expertise en séquençage « lectures longues » avec la technologie Oxford Nanopore.
Les objectifs de la plateforme sont de mettre à disposition des équipes de recherche publiques et privées des outils et des compétences pour mener à bien leurs projets en génomique, transcriptomique et épigénétique. Elle dispose d’instruments de pointe pour le séquençage NGS (Oxford Nanopore et Illumina), le génotypage Crispr-Cas9 à haut débit (HRM) et l’analyse de l’expression génique (qPCR et ddPCR).
La plateforme est définie selon une charte. Elle propose des prestations de services pour la prise en charge d’analyses selon une grille tarifaire, et des collaborations de recherche pour la mise en place de projets avec développement de méthodes spécifiques.
Réseaux
La plateforme est membre du Cortecs , un réseau de plateformes scientifiques de recherche et service de l’université de Strasbourg, en partenariat avec le CNRS et l’INSEM.
La plateforme labellisée IBiSA , un groupement d’intérêt scientifique (GIS) qui mène une politique nationale sélective de labellisation et de soutien aux plateformes de biologie, santé et agronomie et centres de ressources biologiques (CRB).
Moyens et équipements
- Séquençage Oxford Nanopore : PromethION P2 et 2 MinION avec adaptateur Flongle
- Séquençage NGS Illumina : MiSeq
- PCR digitale: Stilla Naica System
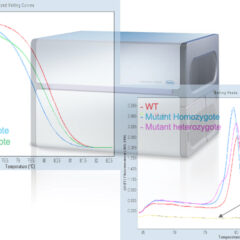
- Analyse d’expression génique par qPCR et génotypage HRM : Roche LC480-II 384 puits
- Contrôle qualité des échantillons et librairies de séquençage NGS : Bioanalyser 2100 (Agilent) ; Qubit (ThermoFisher Scientific)
Comment soumettre un projet à la plateforme?
Pour soumettre un projet à la plateforme, contactez-nous par courriel à l’adresse ibmp-aeg@unistra.fr
Nous vous répondrons dans un délai de 7 jours afin de définir la meilleure stratégie à adopter et l’approche technologique à envisager. Un devis sera ensuite établi. Le délai de réalisation des expériences vous sera communiqué après validation des échantillons. Dans certains cas, un échange avec un bioinformaticien vous sera proposé avant le lancement du projet.