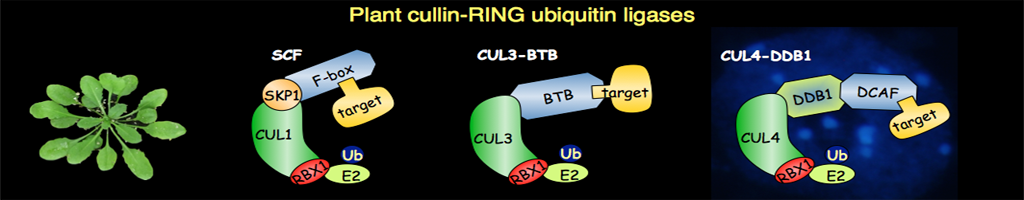
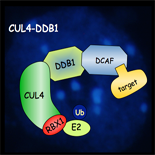
La protéolyse ubiquitine-dépendante est le mécanisme majeur de dégradation sélective des protéines cytoplasmiques et nucléaires dans toutes les cellules eucaryotes. Ce mécanisme joue un rôle essentiel dans la dégradation de protéines régulatrices contrôlant de nombreux processus cellulaires comme le cycle cellulaire et différentes voies de signalisation impliquées dans le développement et la réponse aux hormones. La protéolyse ubiquitine-dépendante se fait en deux étapes, la protéine cible étant tout d’abord modifiée de manière covalente par l’ubiquitine puis dégradée par le complexe protéique 26S protéasome. Certaine protéines ubiquitylées peuvent également être dégradées par un mécanisme d’autophagie sélective. Le transfert de l’ubiquitine sur la protéine cible nécessite des enzymes appelées ubiquitine protéine-ligases (E3) qui déterminent la spécificité de la réaction d’ubiquitylation.
Des résultats récents montrent que ce mécanisme de régulation post-traductionnelle est utilisé de manière prépondérante chez les végétaux supérieurs. En particulier, la voie ubiquitine-protéasome joue un rôle important dans la transduction des signaux endogènes et exogènes comme la lumière ou les phytohormones. Sur la base des génomes d’Arabidopsis et du riz, plus de 1000 enzymes de type E3 ubiquitine protéine-ligases ont été prédites. Cependant, la grande majorité de ces enzymes E3 n’ont pas encore été caractérisées fonctionnellement et leurs substrats restent inconnus. Une meilleure compréhension de ces mécanismes de régulation est importante d’un point de vue académique mais présente également un fort potentiel en biotechnologie et an agronomie.
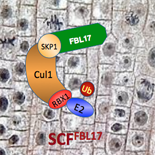
Au laboratoire, nous étudions certaines E3 ubiquitine protéine-ligases impliquées dans le contrôle du cycle cellulaire, la signalisation hormonale et la régulation post-transcriptionnelle (PTGS), chez la plante modèle Arabidopsis thaliana. Afin d’élucider et de caractériser leurs fonctions cellulaires, nous utilisons différentes approches expérimentales comme la génétique, la physiologie moléculaire, la biologie et l’imagerie cellulaire et la biologie structurale.